di Mian Giovanni
Introduzione
La vite (Vitis ssp.) viene coltivata per il consumo umano da circa 7000 anni e poche colture hanno avuto un impatto storico, culturale e sociale come essa.
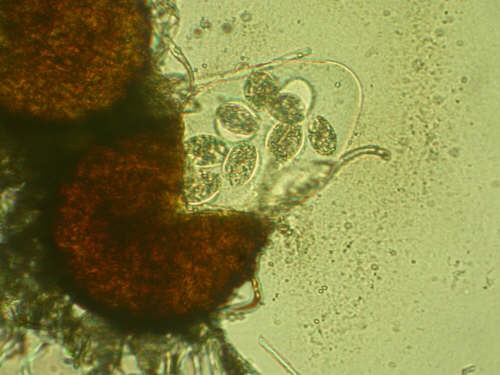
Nel ciclo di produzione fondamentale è il controllo fitosanitario, per preservare sia la produzione sia la qualità delle uve. Il controllo delle patologie, principalmente della peronospora (Fig1) e dell’oidio (Fig. 2), viene attuato principalmente attraverso l’uso di prodotti chimici. Negli ultimi anni, però, si sta cercando di ridurre gli input chimici nella gestione del vigneto per diversi fattori, come i problemi di impatto ambientale, la salute umana ed anche di costi per gli agricoltori. Un metodo sicuro volto a garantire produzioni abbondanti, di qualità e con un minimo uso di agrofarmaci è l’uso di varietà geneticamente resistenti ottenute tramite incrocio.

Fig. 1 Peronospora su foglia di vite – Fig. 2 Oidio su acini di vite
Indipendentemente dal patogeno, esistono 2 strategie che la pianta usa per difendersi dalle infezioni: la resistenza alla penetrazione e la morte cellulare programmata. La resistenza alla penetrazione blocca la breccia che attua il patogeno nella parete e nella membrana cellulare e quindi previene la formazione delle strutture fungine atte a perforare. La morte cellulare programmata è invece esercitata dentro la cellula infettata ed induce la morte della stessa (risposta ipersensibile HR; necrosi), bloccando in tal modo l’apporto di nutrienti richiesti dal fungo per la crescita e lo sviluppo. Il riconoscimento pianta patogeno e la successiva attivazione delle risposte difensive della pianta vengono rappresentati con il così detto “Plant immunity system” (Jones e Dangl 2006) (Fig. 3) dove il riconoscimento intracellulare del patogeno è mediato da una classe di recettori proteici codificati dai geni R che determinano la resistenza a patogeni virali, batterici e fungini oltre che a nematodi e insetti (Martin et al, 2003). Questi sono dei geni conservati nel genoma che rappresentano un’importante arma di difesa che viene attivata quando c’è la necessità di combattere l’invasione dei patogeni. In particolare, nel regno vegetale ogni singola cellula può riconoscere la presenza del patogeno. La maggior parte dei geni R codificano per recettori composti da un sito di legame nucleotidico (NBS) implicato nella trasduzione del segnale e un dominio ricco in ripetizioni di leucina (LRR) implicato nel riconoscimento del patogeno. In particolare, il genoma della vite codifica una grande famiglia di geni NBS-LRR che si trovano raggruppati in ripetizioni a tandem in diverse regioni genetiche (Moroldo et al. 2008, Velasco et al. 2007).
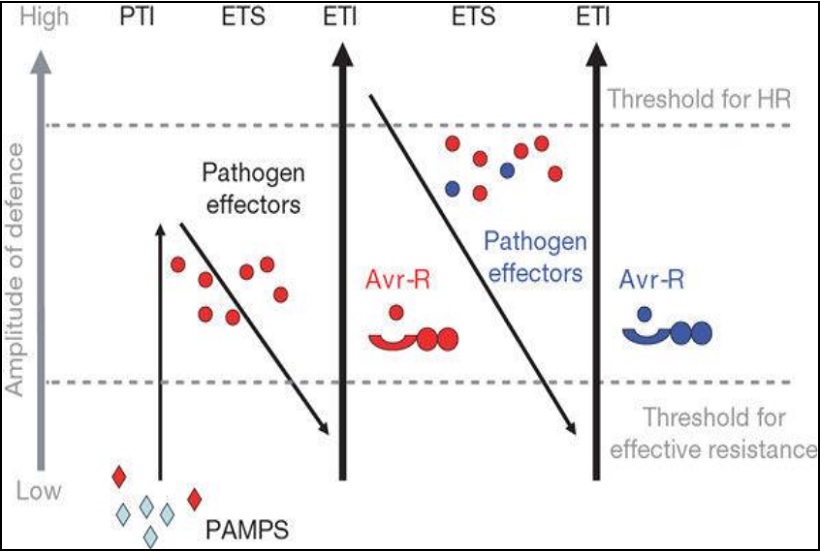
Fig. 3 Sistema immunitario delle piante.
I geni di resistenza si prestano ottimamente per il breeding delle varietà di vite volto ad ottenere resistenze alle malattie; è quindi una strategia diretta per incrementare l’efficienza aziendale e limitare l’impatto ambientale di una costante lotta che prevede l’uso di fungicidi in interventi che possono arrivare anche a 20 in una stagione.
Un altro importante carattere, in particolare per il mercato dell’uva da tavola, è il così detto Seedlessness (apirenia), presente in molte varietà sia per il consumo fresco sia per quello essiccato. Il breeding tradizionale è troppo lento per sopperire alla costante ricerca di nuove varietà con diversi caratteri mentre la selezione attuata con biotecnologie molecolari e genetiche può aiutare enormemente il breeder nell’ottica del miglioramento genetico. In questo aspetto, i marcatori molecolari sono un enorme potenziale per migliorare l’efficienza e la precisione del breeding convenzionale attraverso la così detta “selezione assistita da marcatori molecolari” (MAS: marker assisted selection). Ci sono dunque molte ed importanti applicazioni del MAS per il breeding delle piante; fra queste, in particolare, la possibilità di selezionare in fasi di sviluppo precoci, usando i marcatori molecolari per i tratti d’interesse. Questo permette ai breeders di scartare le piante (selezione negativa) con geni non desiderati e concentrare l’attenzione su un minore numero di individui nelle generazioni successive, senza dimenticare il vantaggio in termini di tempo che si ha selezionando in fasi precoci senza dover aspettare il manifestarsi del carattere. Questo schema è dunque di grande impatto per i programmi di miglioramento delle specie perenni, come la vite, che hanno un lungo periodo di giovanilità, specialmente per i genotipi dove lo screening delle popolazioni può esser fatto solo al manifestarsi del carattere in oggetto in fase adulte (Wan Y et al. 2008). Un marcatore molecolare può essere definito come quel locus genomico, rilevabile con sonde o inneschi (=primer) specifici che, in virtù della sua presenza, contraddistingue in modo caratteristico ed inequivocabile il tratto cromosomico con il quale si identifica e le regioni che lo circondano alle estremità.
Nel caso in cui siano note le sequenze nelle regioni fiancheggianti il marcatore è possibile disegnare primers specifici in grado di amplificare, tramite PCR (Fig. 4), il locus microsatellite. I microsatelliti possono quindi essere rivelati attraverso la costruzione di una biblioteca genomica a piccoli inserti. Alternativamente possono essere usati primers disegnati per specie affini. La tecnica utilizzata per la loro rilevazione è la PCR (Polymerase Chian reaction). Il sequenziamento attraverso PCR porta alla determinazione della sequenza nucleotidica all’interno di un frammento di DNA amplificato attraverso la PCR, utilizzando primers (lunghi 15-35 bp) specifici per un particolare sito genomico (Caramelli, 2009).

Fig. 4 Termociclatore per PCR.
Obiettivo
Obiettivo di questo lavoro è di introgredire in varietà coltivate, agronomicamente valide, geni di resistenza attraverso l’incrocio tra varietà donatrici di geni R e varietà, appunto, già in coltivazione oppure incrociare diverse accessioni di specie resistenti. In questo modo si ottengono degli ibridi che nei successivi anni andranno reincrociati con il parentale agronomicamente valido al fin di recuperare il genoma della buona varietà preservando i geni di resistenza introgressi. Ulteriormente, l’obiettivo è anche di riscontrare diverse resistenze negli individui in modo tale da ottenere la piramidazione dei geni R e quindi ottenere varietà con uno spettro di resistenza più largo e maggiormente difensive. Per questo scopo, si è deciso di attuare incroci tra specie donatrici di geni R e varietà già valide, sia per uva da vino sia per quella da tavola. Successivamente, tramite tecniche biomolecolari inerenti l’estrazione del DNA, l’amplificazione tramite PCR e il sequenziamento di marcatori molecolari si è voluto vedere se la progenie avesse ereditato geni R e se nei singoli individui ci fossero contemporaneamente più resistenze.
Materiali e metodi
Lo studio è iniziato con gli incroci tra le varietà o accessioni indicate in Tabella 1.
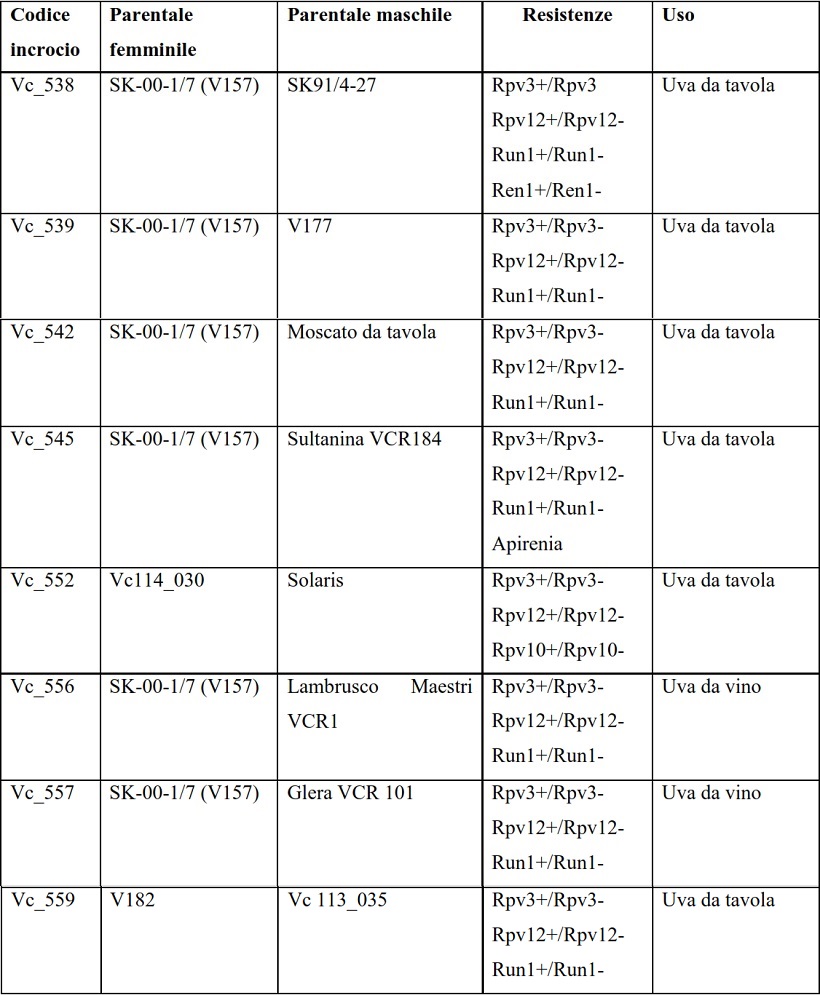
Tabella 1: Incroci effettuati e relative generalità
Dopo gli incroci, al termine della stagione, dai grappoli sono stati estratti i semi e da questi si sono ottenute le nuove piante. Dalle foglie di queste nuove piante, il DNA è stato estratto con il metodo di Doyle e Doyle (1990) e poi è stato quantificato.
Successivamente, per verificare la presenza dei geni in oggetto nel DNA, in primo luogo si sono effettuate delle PCR con i marcatori molecolari per i geni in oggetto, al fine di amplificare i tratti d’interessi. Successivamente, dal prodotto della PCR si è passati al sequenziamento dei frammenti allelici tramite elettroforesi capillare.
Risultati
Per ogni incrocio vengono riportate delle tabelle in cui sono indicati, per ogni gene e per ogni combinazione di geni, il numero di individui ottenuti sul totale della progenie.
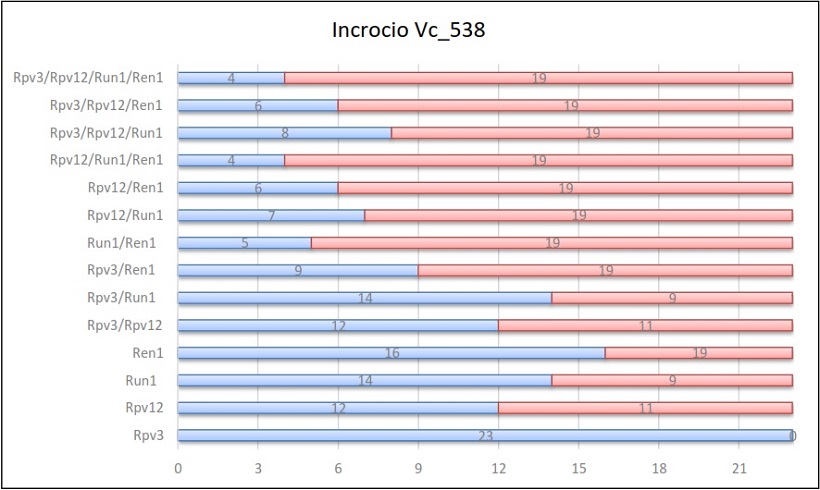
Risultati dell’incrocio 538
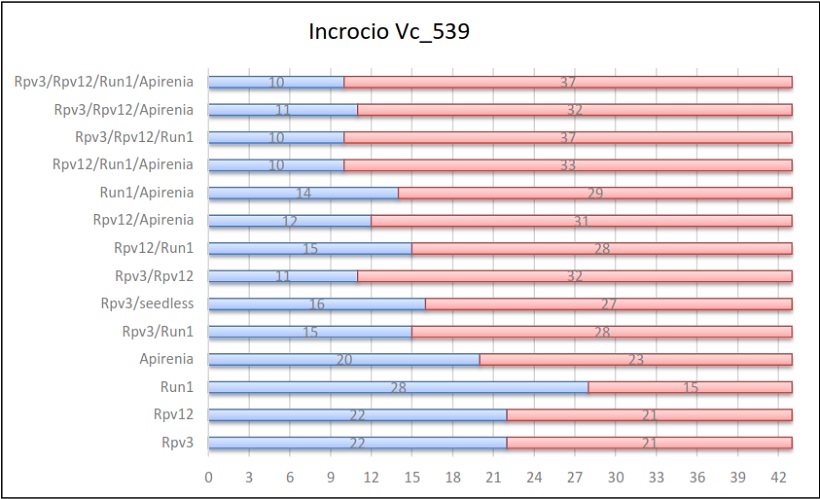
Risultati dell’incrocio 539
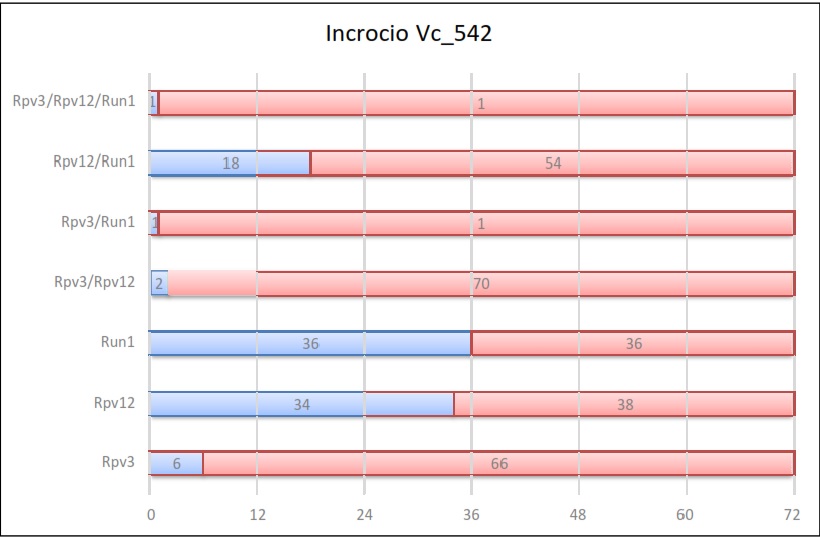
Risultati dell’incrocio 542
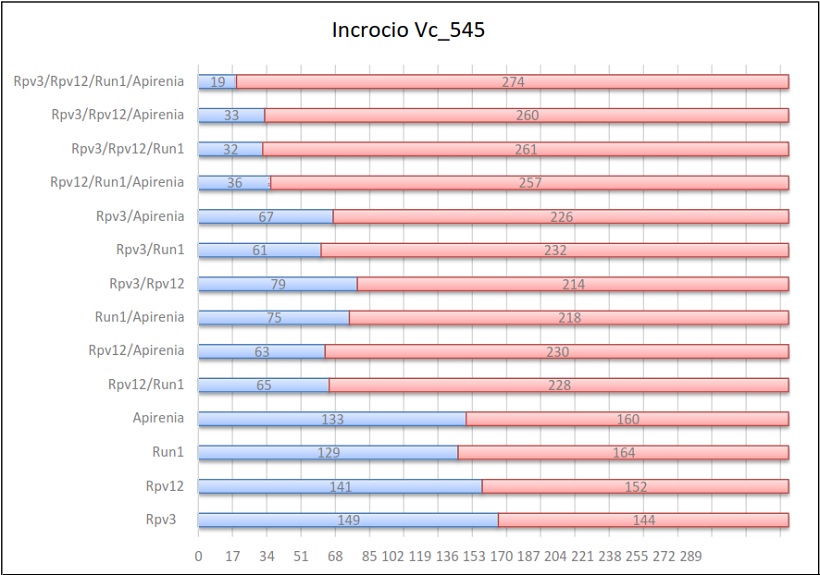
Risultati dell’incrocio 545
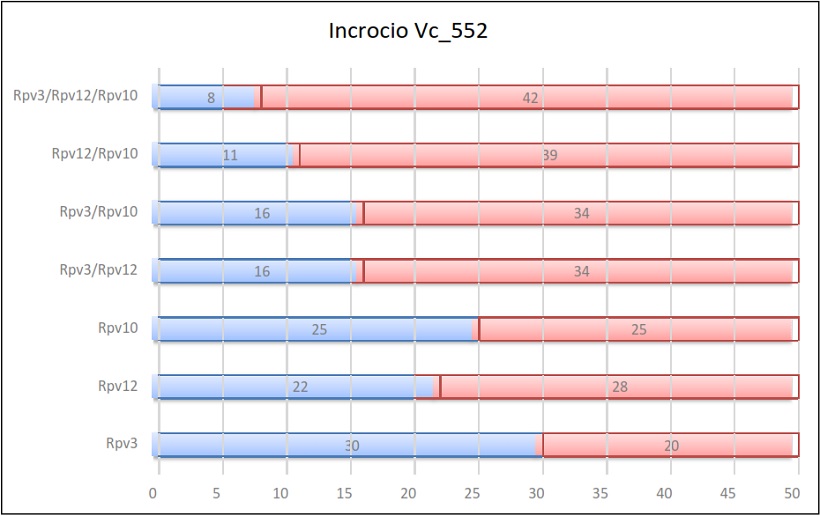
Risultati dell’incrocio 552
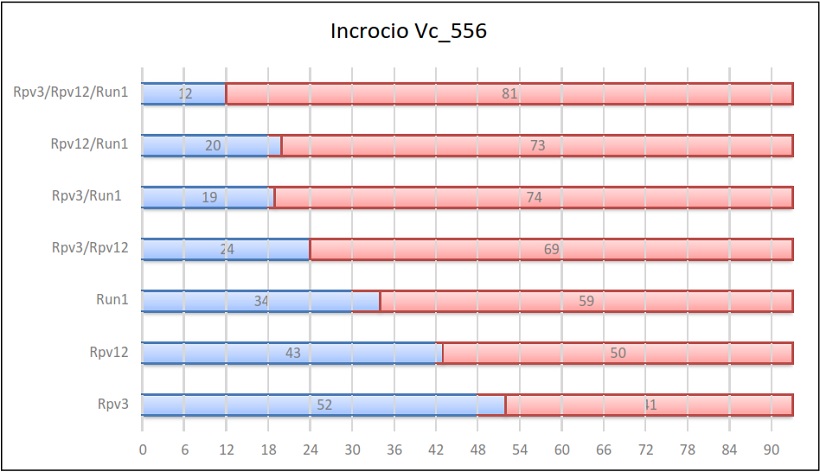
Risultati dell’incrocio 556
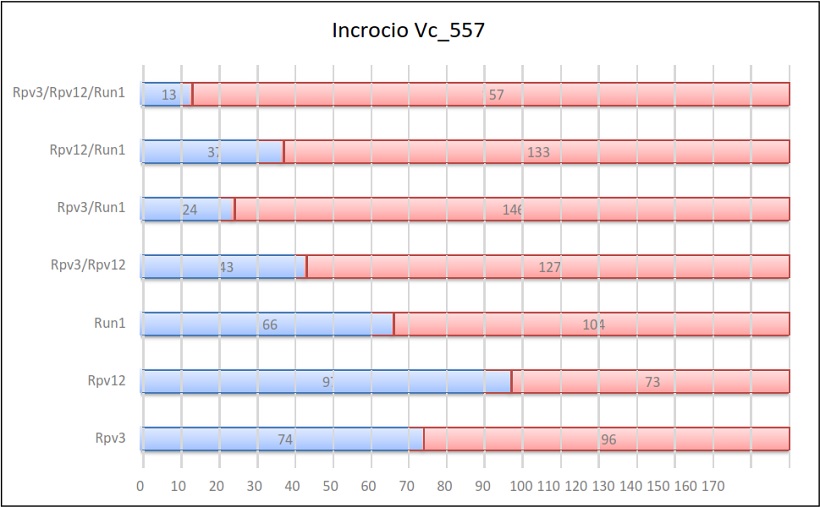
Risultati dell’incrocio 557
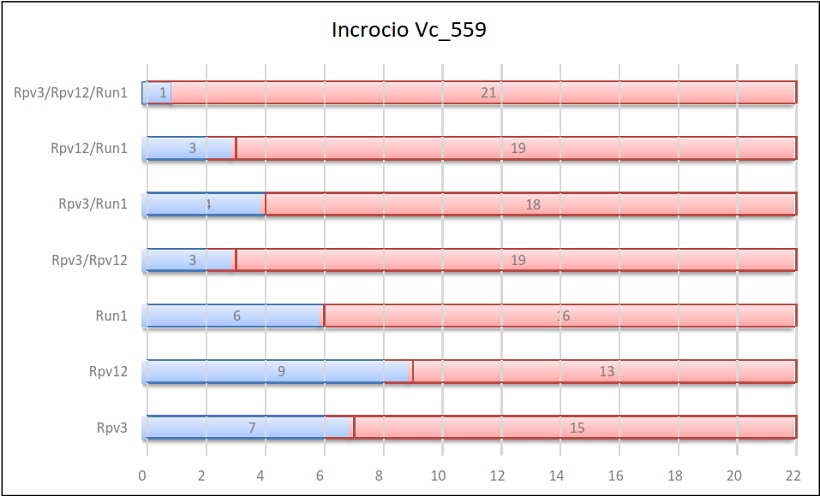
Risultati dell’incrocio 559
Confronto della percentuale di ereditarietà dei geni Rpv3, Rpv12 e Run1
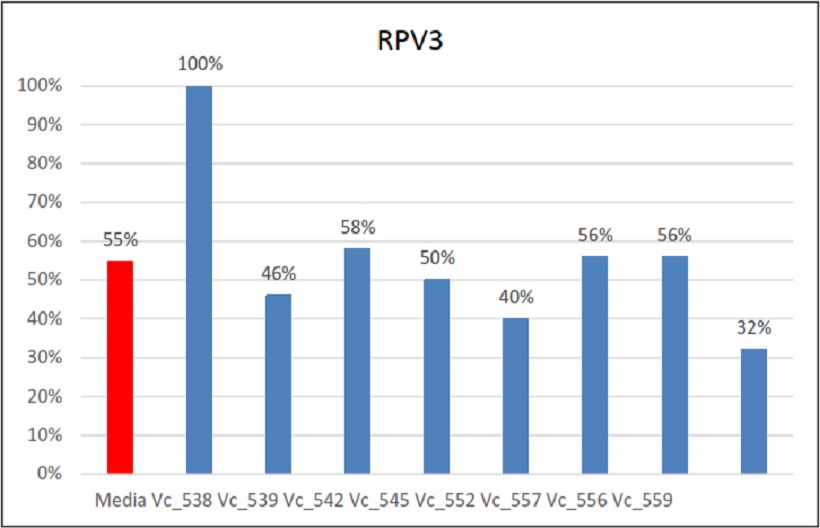
Percentuali di ereditarietà e relativa media di Rpv3
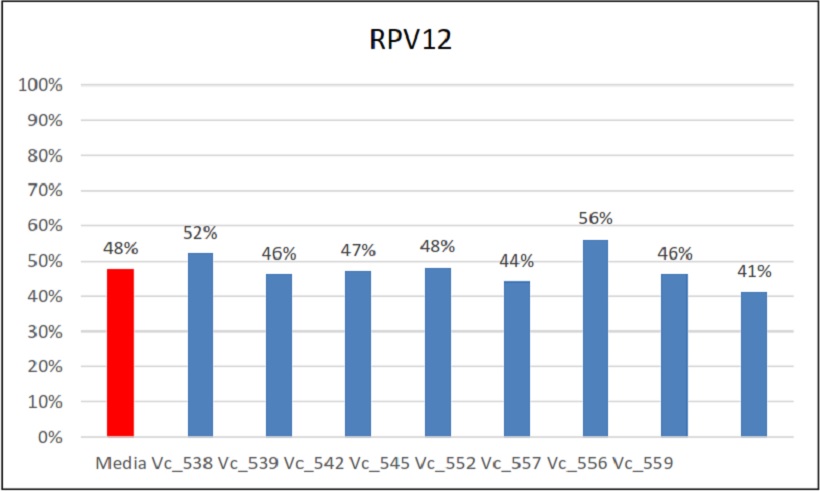
Percentuali di ereditarietà e relativa media di Rpv12
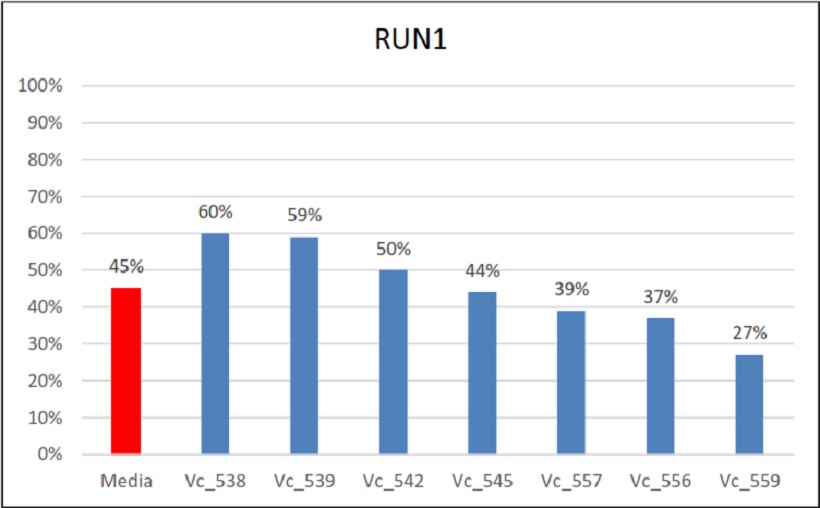
Fig. 11 Percentuali di ereditarietà e relativa media di Run1
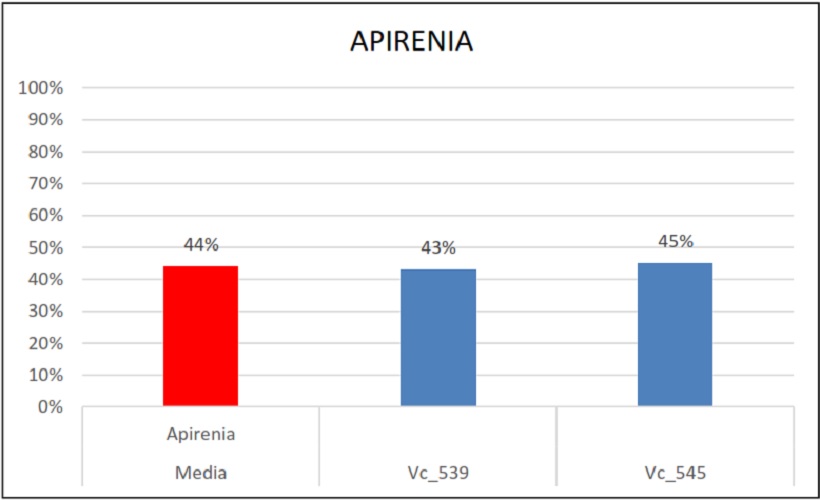
Percentuali di ereditarietà e relativa media dell’apirenia
Discussione
Ad oggi, in viticoltura, vengono usati il 67 % di tutti i fungicidi utilizzati in agricoltura, ricordando che la superficie occupata dalla vite è pari solo al 3,3 % di tutta la superficie agricola mondiale (Eurostat, 2007). La coltivazione della vite quindi, specialmente per quanto concerne la protezione contro le malattie, risulta delicata. Questo enorme uso di composti chimici apporta numerose problematiche, dal punto di vista ambientale ma anche da quello della salute umana, dei microrganismi, dei residui e del pericolo di selezione di ceppi resistenti dei patogeni (Quaglia et al. 2011).
In base a queste motivazioni l’obiettivo di questa tesi è stato quello di ottenere varietà resistenti e con caratteri importanti per il mercato che quindi necessitino di un uso minimo di fungicidi. A tal fine si sono ibridate varietà già coltivate con varietà portanti nel proprio genoma i geni di resistenza alle malattie oppure si sono incrociate 2 varietà resistenti per ottenere individui con più geni piramidati in un solo individuo. In particolare il più alto obiettivo è quello di ottenere varietà con contemporaneamente più geni di resistenza in modo che la futura varietà presenti diverse resistenze allo stesso patogeno e resistenze contro diversi patogeni. In alcuni casi si è anche voluto unire queste resistenze a caratteri merceologici importanti, come ad esempio l’apirenia per quanto concerne l’uva da tavola.
Si sono quindi eseguiti gli incroci nel 2016 tra questi tipi di varietà, nella azienda agraria di dell’Università di Udine “Antonio Servadei” e presso la sezione sperimentale dei Vivai cooperativi di Rauscedo “Casa 40”. Dopo l’incrocio, una volta ottenuti i grappoli, si sono estratti i semi e da questi, nell’aprile seguente, sono nate le piantine che rappresentano gli ibridi frutto dei diversi incroci. A questo punto si è proseguito con la selezione assistita tramite l’uso di marcatori molecolari: il Dna è stato estratto e quantificato e successivamente con specifici primer è stata eseguita la Pcr Touchdown al fine di amplificare solo le sequenze d’interesse. In fine si è sequenziata la presenza dei marcatori collegati al gene d’interesse tramite elettroforesi capillare. I dati sono stati corretti ed elaborati e si sono ottenuti i risultati finali.
In particolare, come marcatore del gene Rpv3 inizialmente era stato utilizzato il marcatore UDV737. Questo marcatore era però omozigote per l’allele resistente (avevano la stessa lunghezza in basi) e nei diversi incroci anche i parentali non resistenti mostravano lo stesso allele non resistente della stesa lunghezza. Non era quindi possibile applicare una selezione in quanto, dalle chiamate alleliche effettuate con software geneMapper dopo elettroforesi capillare, non si potevano distinguere questi alleli nelle diverse combinazioni alleliche. Si sarebbe dovuto aspettare la fenotipizzazione. Si è deciso, quindi, di usare un altro marcatore, UDV305, che ha presentato lunghezze alleliche in paia di base diverse e quindi si sono potuti distinguere con facilità gli individui che avevano ereditato l’allele resistente da quelli che invece non l’avevano ereditato. A questo punto, avendo usato i 2 marcatori per Rpv3, di cui uno e localizzato a monte del gene e l’altro a valle, si poteva anche distinguere le combinazioni alleliche precedentemente ottenute con UDV737 effettuando un controllo incrociato. Questa casistica di utilizzo di 2 marcatori, uno a monte e uno a valle, è anche la migliore delle analisi. Infatti, più il marcatore è lontano dalla regione di interesse, maggiore è le probabilità che avvenga una ricombinazione. In particolare la probabilità è del 1% per ogni cM (centi Morgan) di distanza (Barcaccia e Falcinelli, 2006). Può quindi avvenire il caso in qui venga sequenziato ed amplificato il microsatellite marcatore, perché presente, ma che in realtà non ci sia il gene d’interesse perché ha ricombinato durante il crossing over. Una casistica simile è stata riscontrata anche per il marcatore per il gene dell’apirenia ma in questo caso, non avendo a disposizione un altro marcatore, per applicare la selezione si sono dovute scartare le combinazioni alleliche in qui non era possibile identificare quello portante il tratto apirenico. Per far ciò si sarebbe dovuto utilizzare, anche qui, un marcatore diverso oppure avere dati della fenotipizzazione per poter capire chi l’avesse ereditato.
Per ogni famiglia analizzata un grande numero di individui nella progenie ha ereditato i geni di resistenza. Questo significa che l’ereditabilità dei suddetti geni risulta essere elevata.
Il primo gene esaminato è stato Rpv3: la media della sua ereditabilità è stata del 55%, un dato che nei relativi incroci non varia di molto. Si è riscontrato però un caso eccezionale, quello dell’incrocio Vc_538. In questo caso tutti gli individui hanno ereditato questo gene di resistenza alla peronospora. Dato particolare, in quanto la segregazione che ci si aspettava era 1:1. Questo può esser verosimilmente spiegato con un’ipotesi di casualità del dato. Probabilmente avendo avuto a disposizione più individui di quelli ottenuti l’ereditabilità di Rpv3 sarebbe cambiata. Ad ogni modo il risultato della fenotipizzazione ha confermato in parte quanto riportato dall’analisi molecolare, in quanto si sono trovati 4 individui che manifestavano sintomi. Questi sintomi però non sono stati chiari. Infatti su questi 4 individui è stata trovata peronospora a mosaico e non macchie d’olio classiche con sporulazione nella pagina inferiore. Verosimilmente, ci può esser stata una iniziale infezione successivamente bloccata dalle resistenze ereditate.
Nel caso del secondo gene di resistenza alla peronospora, Rpv12, quest’ultimo ha mostrato un’eredità del 48%. Nei singoli incroci i valori della singola ereditabilità in quella combinazione infatti non si discosta molto da questa media.
Per il gene di resistenza all’oidio Run1, la media di tutti gli incroci è stata del 45%. Anche in questo caso valori simili si sono ottenuti per ogni incrocio con una maggiore ereditabilità nell’incrocio Vc_538 (60%) e Vc_539 (65%).
In alcuni casi è stato preso in considerazione negli incroci anche il gene Ren1 (Vc_538) che ha mostrato un’ereditabilità del 70% e Rpv10 (Vc_552) che ha mostrato un’ereditabilità del 50%. Nello specifico, Ren1 ha mostrato un’ereditabilità del 25% più alta dell’altro gene di resistenza Run1 (45%) mentre per Rpv10 si è notata un’ereditabilità minore del 5% rispetto Rpv3 (55%) e del 2% maggiore rispetto Rpv12 (48%).
Ulteriormente, il gene portante il carattere dell’apirenia è stato usato in 2 popolazioni: nella prima è stato ereditato dal 45% (Vc_545) della progenie e nella seconda dal 43% (Vc_539). La media è risultata esser del 43%, un dato che non si discosta di molto da quello individuato da uno studio precedente condotto da C. Bergamini et al. (2013) in qui videro che l’ereditabilità di questo gene si attestava al 49%.
Oltre al fatto dell’ottimo risultato nel aver ottenuto molti individui resistenti in tutti gli incroci effettuati, un aspetto molto importante è che per ogni incrocio si siano ottenuti un buon numero di individui portanti diverse combinazioni di geni di resistenza ed altrettanti che hanno ereditato tutti i geni presenti nel singolo incrocio. In particolare questi sono i risultati che più vanno presi in considerazione. Ottenere varietà con piramidate più resistenze verso lo stesso patogeno e contemporaneamente più resistenze contro diversi patogeni è il risultato migliore che si possa avere. Questi individui infatti presenteranno un elevato grado di resistenza e sarà più difficile per il patogeno riuscire a superare queste barriere evolvendo tramite la selezione naturale. Questi individui con diverse resistenze piramidate sono la base di partenza per continuare con il miglioramento genetico anche per ulteriori caratteri. In questo lavoro, si sono ottenuti anche individui che portassero 3 geni di resistenza (fra peronospora e oidio, in diverse combinazioni) e il tratto dell’apirenia.
L’utilizzo delle conoscenze della genetica e le moderne tecniche molecolari per ottenere varietà migliorate, come per le resistenze alle malattie, sono, quindi, i mezzi più efficaci che abbiamo a disposizione per ottenere una riduzione drastica nell’uso dell’agro chimica. Inoltre, possiamo ipotizzare di associare ai caratteri di resistenza altri caratteri per molti tratti importanti. In questo modo, coltivatori, consumatori e ambiente potranno trarre indubbi vantaggi in termini di salute pubblica e conservazione dell’ecosistema in un’ottica di sviluppo sostenibile.
Bibliografia
- Caramelli David, Antropologia molecolare, Firenze University Press, 2009
- EUROSTAT EC. The use of plant protection products in the European Union. Data 1992- 2003. Luxembourg: Office for Official Publications of the European Communities, 2007. ISBN 92-79-03890-7.
- Bergamini et al, 2012. Validation assay of p3_VvAGL11 marker in a wide range of genetic background for early selection of stenospermocarpy in Vitis vinifera L
- Jones JD, Dangl JL (2006) The plant immune system. Nature 444:323Kast WK, Stark-Urnau M, Seidel M, Gemmrich AR (2001) Interisolate variation of virulence of Plasmopara viticola on resistant vine varieties. Bull OILB/SROP 24:45–49
- Moroldo M, PaillardS,Marconi R,FabriceL,Canaguier A,Cruaud C etal. A physical map of the heterozygous grapevine ‘Cabernet Sauvignon’ allows mapping candidate genes for disease resistance. BMC Plant Biol 2008; 8: 66. 31
- Velasco R, Zharkikh A, Troggio M, Cartwright D, Cestaro A, Pruss D et al. A high quality draft consensus sequence of the genome of a heterozygous grapevine variety. PLoS ONE 2007; 2:
- Wan Y, Schwaninger H, He P, Wang Y. Comparison of resistance to powdery mildew and downy mildew in Chinese wild grapes. Vitis. 2007;46:132.
Sitografia
Giovanni Mian si è diplomato all’Istituto tecnico agrario “P. d’Aquileia” di Cividale del Friuli Ha conseguito la laurea triennale in Scienze agrarie presso l’Università degli studi di Udine. Successivamente si è iscritto, nello stesso ateneo, al corso di laurea magistrale in Scienze e tecnologie agrarie, laureandosi ad ottobre del 2017 con il massimo dei voti. Vanta numerose esperienze lavorative nel settore agricolo e nel settore della sperimentazione di molecole usate come fitofarmaci. E-mail: giovanni.mian@outlook.it