Analisi della sintesi di etilene in una popolazione segregante di melo e caratterizzazione molecolare di geni candidati
di Daniele Puliti
Introduzione
Il melo è una pianta da frutto originaria della zona caucasica e, con una superficie di oltre 5 milioni di ettari e una produzione di oltre 89 milioni di tonnellate, è la specie da frutto più coltivata al mondo (dati FAO). La specie coltivata prende il nome di Malus domestica e fa parte della famiglia delle Rosacee, sottofamiglia Maloidee. Dal punto di vista genetico, recenti studi hanno evidenziato che il genoma si sia originato dalla autopoliploidizzazione o dell’ibridazione tra due membri dello stesso taxa con x=9, seguiti da diploidizzazione e anaeploidizzazione a x=17.
La grande variabilità della specie è dovuta sia alle numerose varietà sia al notevole numero di cloni. I campioni analizzati in questo lavoro appartengono a piante derivate dall’incrocio di due cultivar molto diffuse ed apprezzate: ‘Fuji’ e ‘Mondial Gala’.
- ‘Fuji’ nasce in Giappone e deriva dall’incrocio di ‘Ralls Janet’ x ‘Delicious’ e presenta una polpa croccante, mediamente zuccherina e poco acidula. Viene raccolta in ottobre e la sua lenta maturazione le permette di mantenere una certa durezza e una lunga shelf life.
- ‘Mondial Gala’ nasce in Nuova Zelanda e deriva dall’incrocio di ‘Kidd’s Orange’ x ‘Golden Delicious’ e presenta una polpa soda, zuccherina e poco acidula. La raccolta avviene in agosto, ha un alto tasso di maturazione e una conservazione minore rispetto a ‘Fuji’.
La maturazione coincide con la fase finale della distensione cellulare del frutto e comprende processi chimico-fisici che aumentano il grado di appetibilità. Nei frutti climaterici la decrescita naturale della respirazione subisce un’inversione, raggiunge un picco per poi decrescere di nuovo. L’aumento della respirazione coincide con un’ulteriore sintesi di etilene, come si può vedere nel riquadro evidenziato del grafico. Al raggiungimento del plateau della curva di maturazione, si ha il picco di etilene e respirazione.
L’etilene è un alchene, avente formula C2H4, che nel frutto in maturazione stimola l’attivazione di enzimi quali perossidasi, amilasi, pectasi, eccetera.
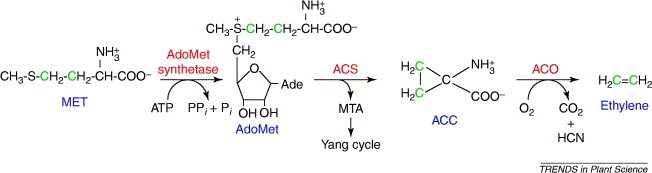
Figura 1 Ciclio dell’etilene, Chae and Kieber, Eto Brute? Role of ACS turnover in regulating ethylene biosynthesis.
Come si può vedere dall’immagine (figura 1), esso è sintetizzato a partire dalla metionina, che si converte in S-adenosil-L-metionina. A questo punto intervengono due enzimi.
- L’acido amminociclopropano carbossilato sintasi (ACS), che catalizza la conversione della SAM in acido amminociclopropano carbossilato.
- L’acido amminociclopropano carbossilato ossidasi (ACO), che catalizza la conversione di ACC in etilene.
Il ruolo delle isoforme di ACS e di ACO1 nella popolazione F x MG è già stato indagato in un precedente lavoro di tesi e non spiega la variabilità fenotipica tra i parentali.
Scopo della tesi
Lo scopo principale di questa ricerca consiste nel caratterizzare il processo di biosintesi di etilene in ‘Fuji’ e ‘Mondial Gala’. Sui frutti raccolti sono stati determinati, in semenzali derivati dall’incrocio di ‘Fuji’ x ‘Mondial Gala’, la data di maturazione e il contenuto di etilene attraverso gascromatografia sui frutti. Questi dati sono stati utilizzati per una analisi QTL allo scopo di identificare le regioni del genoma del melo coinvolte nella determinazione della data di maturazione e del contenuto di etilene. Le regioni genomiche interessate dalla presenza di QTL saranno inoltre analizzate per identificare geni putativamente in grado di modulare il processo di maturazione, localizzati nella regione del QTL. Su questi geni è stato verificato il livello di espressione con PCR quantitativa sul cDNA di frutti di ‘Fuji’ e ‘Mondial Gala’ in quattro diversi momenti di conservazione: alla raccolta e dopo 7, 14, 21 giorni.
Materiali e metodi
Lo studio è stato effettuato su una popolazione di semenzali (di cui 194 utilizzati per il mappaggio) ottenuti dall’incrocio di ‘Fuji’ x ‘Mondial Gala’. campioni sono stati raccolti e pesati, stoccati per due settimane e analizzati al gascromatografo.
Sulla sequenza genomica del melo sono stati identificati il gene ACO 1 e le sue isoforme ACO 1bis, ACO 2, ACO 3, ACO 3bis. Su questi geni sono stati disegnati i primer ed è stata effettuata una PCR per amplificarli. Il prodotto è stato in seguito purificato e sequenziato.
Sul gene ACO 1bis è stata svolta una TSP al fine di analizzare gli SNP identificati col sequenziamento. In questa tecnica vengo utilizzati tre primer: un primer forward locus specifico, un primer reverse locus specifico e un primer allele specifico che possiede una coda di basi arbitrarie.
Sui cDNA di frutto di ‘Fuji’ e ‘Mondial Gala’ è stata svolta un’analisi PCR dell’espressione del gene ACO 1bis con i primer TSP alleli specifici (quindi per entrambi gli alleli).
La banda dell’allele specifico è stata individuata sui 194 genotipi della popolazione al fine di mappare il gene ACO 1bis su ‘Mondial Gala’, utilizzando i dati di genotipizzazione della popolazione FJ x MG derivanti dallo SNP Chip da 20K.
L’analisi QTL è stata svolta per il contenuto di etilene sui frutti ed epoca di maturazione con test di Kruskal-Wallis ed Interval Mapping.
Inoltre, sono stati individuati altri geni che potrebbero influire sulla sintesi di etilene nelle regioni dei QTL in cui è presente l’isoforma ACO studiata. È stata quindi studiata l’espressione dei geni candidati tramite PCR quantitativa.
Risultati
Raccolta e analisi dei campioni
La raccolta dei frutti è stata eseguita seguendo il parametro dell’osservazione del colore di fondo.
Come si può vedere dai grafici (figura 2), che riportano la percentuale dei campioni divisi in base al contenuto di etilene e ai giorni dall’inizio dell’anno, le analisi dei campioni al gascromatografo e le date di maturazione evidenziano una distribuzione dei dati bimodale.
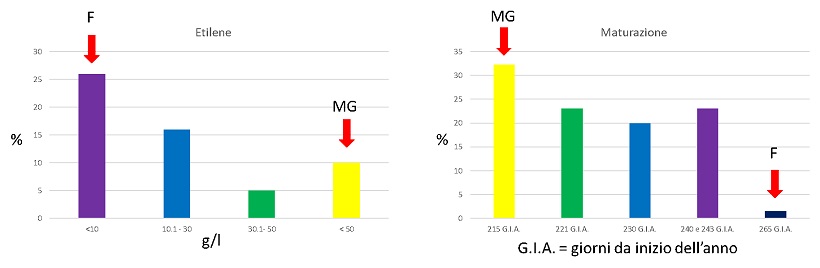
Figura 2 Distribuzione dei campioni in base al contenut di etilene e alla data di maturazione
Come previsto, i parentali si trovano agli estremi della variabilità evidenziata dai semenzali della popolazione.
Amplificazione delle isoforme di aco
Si è proceduto all’identificazione delle popolazioni dei geni ACO annotati grazie al network genomico Phytozome 12 e il Dataset di Genomica delle Rosacee.
Come prima caratterizzazione si è proceduto all’allineamento delle sequenze con il software Clustal omega dei cinque membri noti della famiglia ACO, al fine di determinare le regioni univoche per disegnare i primer e per verificare il livello di omologia tra i geni: come si può notare dall’albero filogenetico (figura 3), ACO 1 e ACO 1bis e ACO 3 e ACO 3bis sono posizionati su cromosomi omeologhi e quindi hanno una omologia di struttura.

Figura 3 Albero filogenetico dei geni analizzati
Sulle sequenze note sono stati disegnati primer con l’ausilio del software Primer3. Nonostante il sito avesse dato condizioni precise di amplificazioni PCR, i geni non sono stati amplificati molto bene e si è dovuti ricorrere ad ottimizzazioni del protocollo di amplificazione correggendo la concentrazione di magnesio e la temperatura di annealing (figura 4).
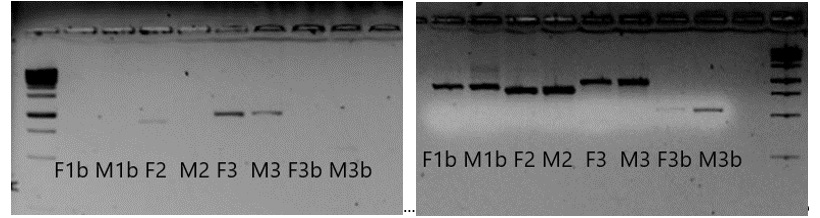
Figura 4 Gel ottenuto dall’amplificazione dei campioni e sua ottimizzazione
Sequenziamento delle isoforme di aco
Una volta ottenute le sequenze, si è osservato che l’isoforma ACO 1bis presentava polimorfismi tra i parentali.
Analizzando la sequenza di ACO 1bis, sono stati trovati SNP e INDEL che rendono polimorfici gli alleli di ‘Fuji’ e ‘Mondial Gala’. Nell’esempio vediamo uno SNP presente in ‘Mondial Gala’ (figura 5).
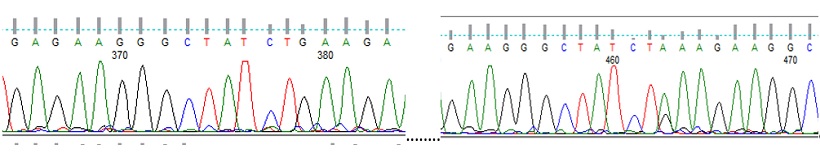
Figura 5 SNP rappresentato da una guanina in ‘Fuji’ e una adenina in ‘Mondial Gala’
Tuttavia, nelle altre isoforme non sono stati trovati polimorfismi di sequenza.
Analisi di espressione di aco1bis in fuji e mondial gala
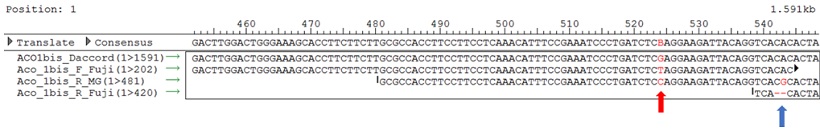
Figura 6 Stop codon e INDEL presenti nella sequenza di ‘Fuji’
Come si può notare invece da quest’altra figura (figura 6), ‘Fuji’ presenta uno stop codon anticipato, rappresentato dalla tripletta TAG, indicato con la freccia rossa, e un INDEL subito a valle, che provoca un frame shift della proteina, indicato con la freccia blu. A causa di questi due fattori, il gene potrebbe essere inattivato, rendendolo incapace di produrre una proteina funzionante.
Il primer allele specifico in grado di genotipizzare anche l’allele di ‘Fuji’ è stato utilizzato per verificare la presenza di trascritti per ACO 1bis in un pool di cDNA di frutto a diversi tempi di conservazione. L’analisi dimostra che il gene è espresso sia in ‘Fuji’ che ‘Mondial Gala’ (figura 7). Il gene quindi è trascritto, ma la proteina non è funzionante, quindi l’analisi di espressione non può spiegare le differenze fenotipiche dei parentali.
Analisi TSP sul gene aco1bis
Lo SNP trovato permette la genotipizzazione degli alleli di ‘Mondial Gala’. Come si può notare dal gel (figura 8), si è ottenuta un’amplificazione molto chiara dell’allele di basso peso molecolare di ‘Mondial Gala’. Grazie alla TSP è stato possibile evidenziare il polimorfismo tra ‘Fuji’ e ‘Mondial Gala’.
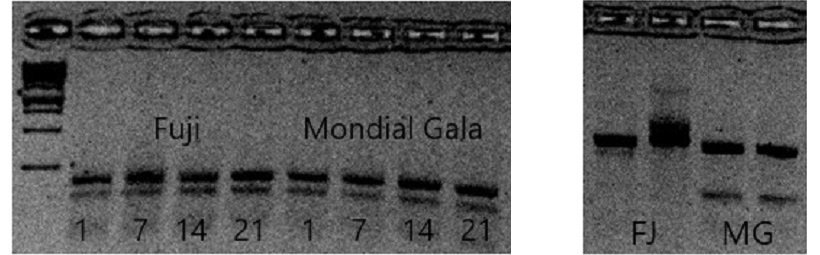
Figura 7 Analisi di espressione del gene ACO1bis nei parentali- Figura 8 SNP evidenziato dal gel
Si è poi proceduto all’analisi del marcatore sui 194 genotipi della popolazione in funzione del mappaggio. Ai campioni che presentavano la banda di basso peso molecolare è stato assegnato il genotipo np, mentre ai campioni che presentavano solo la banda locus specifica è stato assegnato il genotipo nn.
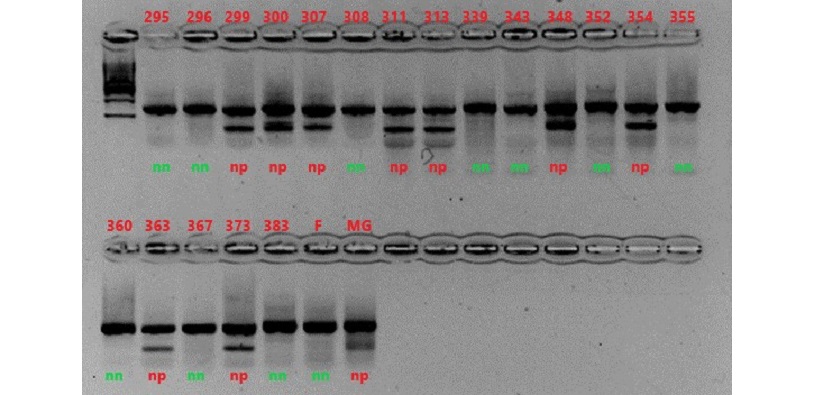
Figura 9 Analisi del marcatore su tutti i genotipi della popolazione
Mappaggio di ACO1bis sul LG5 di ‘Mondial Gala’
Il gene ACO 1bis è stato mappato su ‘Mondial Gala’ nel LG 5. Come si può notare dalla mappa (figura 10), il gene, evidenziato in rosso si trova in fondo al cromosoma.

Figura 10 Mappa del LG5 su ‘Mondial Gala’
QTL per la data di maturazione sul LG 5
Dalla curva di interval mapping (figura 11) si può notare come ACO 1bis sia sotto la soglia di LOD (misurata con il test di permutazione) e fuori dal QTL, mentre sono stati identificati tre geni al di sopra di questa soglia, che sono RmlC like cupin, subtilasi e germin like protein. Questi, in altri frutti sono stati associati alla senescenza e alla maturazione del frutto.
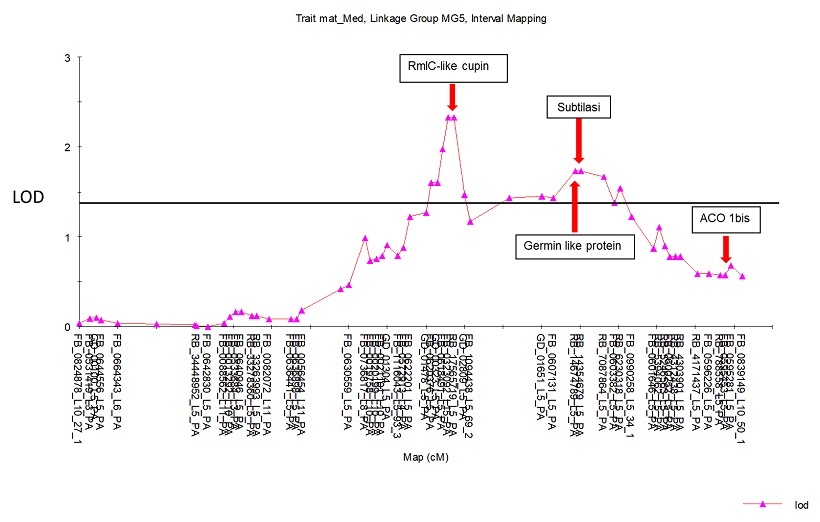
Figura 11 Curva di Interval Mapping sul LG5
Si evince quindi che il gene ACO 1bis non concorre ad influire sul carattere quantitativo di produzione di etilene.
Analisi QTL
I geni candidati mostrati nella slide precedente sono stati trovati in altri linkage group, in particolare l’11 e il 13.
Tramite il programma MapQTL 4.0 sono stati identificati QTL associati all’epoca di maturazione e al contenuto di etilene, in particolare due QTL nei LG 8 e 14 per il contenuto di etilene e sei QTL per la data di maturazione (LG 2, 5, 6, 11, 13, 14) (figura 12).

Figura 12 Tabella derivata dall’analisi QTL della data di maturazione
In un recente lavoro di ricerca, Urrestarazu et al. hanno descritto diversi QTL associati all’epoca di maturazione che si trovano nello stesso linkage group di alcuni QTL identificati in questo lavoro di tesi, ma che non si trovano nelle stesse posizioni. Bisogna però tenere conto del diverso approccio utilizzato, che potrebbe influire sulla identificazione dei QTL: infatti, mentre in questa tesi si è lavorato su una popolazione segregante, Urrestarazu et al. hanno lavorato su germoplasma di melo.
Nei linkage group studiati sono stati identificati altri geni candidati che possono influenzare la maturazione.
PCR real time di geni candidati
I tre geni sono espressi a livelli molto inferiori rispetto all’house keeping actina, però subtilasi, GLP e RmlC sono enzimi, quindi anche in basse quantità potrebbero comunque influire sensibilmente sull’espressione del carattere.
Questi tre geni sono stati analizzati con la PCR real time, utilizzando gli stessi pool di cDNA usati per le analisi di espressione su ACO 1bis (figura 13).
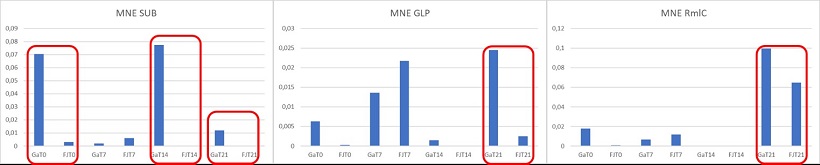
Figura 13 Risultati dell’analisi PCR quantitativa dei geni candidati
La subtilasi presenta un’espressione differenziale tra ‘Mondial Gala’ e ‘Fuji’. Questo gene va ad influire la maturazione di alcune specie di interesse come papaya, melone e vite, e potrebbe influire sulla produzione di etilene in melo.
La GLP presenta differenze marcate tra le due cultivar solo dopo 21 giorni di conservazione.
La RmlC presenta livelli di espressione relativamente più alti dopo 21 giorni di conservazione con scarse differenze fra le due cultivar. Inoltre, siccome il picco di etilene si ha al 14° giorno di conservazione, un’espressione del gene al 21° giorno risulta tardiva.
Conclusioni
Dai risultati ottenuti, si possono trarre le seguenti conclusioni:
- L’analisi al gascromatografo ha confermato la differenza nella produzione di Etilene fra ‘Mondial Gala’ e ‘Fuji’.
- I caratteri «produzione di etilene» ed «epoca di maturazione» segregano nella popolazione in esame.
- L’analisi di sequenza dei geni ACO analizzati ha evidenziato solo per ACO 1 bis sul cromosoma 5 polimorfismi di sequenza fra ‘Fuji’ e ‘Mondial Gala’.
- L’analisi di sequenza del gene completo ha rivelato che gli alleli di ACO 1 bis di ‘‘Mondial Gala’’ sono perfettamente funzionanti, mentre in ‘‘Fuji’’ sono stati identificati due polimorfismi (uno SNP e un INDEL) che possono annullarne l’attività.
- La presenza dei trascritti di ACO 1bis in ‘‘Fuji’’ non può spiegare l’effetto sul fenotipo.
- L’analisi QTL ha identificato in ‘Mondial Gala’ regioni associate sia per il contenuto di etilene sia per l’epoca di maturazione.
- L’isoforma ACO 1bis si è localizzata fuori dalla regione del QTL sul LG5.
- La subtilasi sembra essere, tra i geni che possono spiegare il QTL sul linkage group 5, il migliore candidato al ruolo di modulatore della maturazione nel frutto di melo.
I risultati ottenuti da questo lavoro sono d’aiuto sia per la comprensione di aspetti ancora oscuri della regolazione genica durante la maturazione sia per lo sviluppo di marcatori molecolari per la selezione assistita.
Sintesi della Tesi di Laurea Magistrale di Daniele Puliti, Relatore prof. Luca Dondini.
Daniele Puliti, diplomato al Liceo Scientifico Ernesto Balducci di Pontassieve, ha conseguito la Laurea Triennale in Scienze Agrarie presso l’Università degli Studi di Firenze e la Laurea Magistrale in Scienze e Tecnologie Agrarie presso l’Università degli Studi di Bologna. E-mail: danipul21@gmail.com